ヒメツリガネゴケのミトコンドリアゲノムの解読
The Mitochondrial Genome of the Moss Physcomitrella patens Sheds New Light on Mitochondrial Evolution
in Land Plants
Kimihiro Terasawa, Masaki Odahara, Yukihiro Kabeya, Tatsuhiko Kikugawa,
Yasuhiko Sekine, Makoto Fujiwara, and Naoki Sato
Molecular
Biology and Evolution (2006) online (2007) in press
doi:10.1093/molbev/msl198
序
これまで植物のミトコンドリアゲノムの解読は,被子植物と緑藻については多数の生物について進められているが,コケ植物では,タイ類のゼニゴケについて報
告されているのみであった。
今回,セン類のヒメツリガネゴケのミトコンドリアゲノムを解読し,さまざまな植物・藻類のミトコンドリアゲノムと比較を行った結果,ゼニゴケや車軸藻類と
きわめてよく似ていることがわかった。
配列の決定
配列決定の詳細は論文を見てください。約半分の50 kbpに関しては,long
PCRによって得られた産物のシークエンシングを行った。さらに,全体のショットガンシークエンシングを行い,アセンブルした。いくつかのギャップを
PCRで埋めて全体を完成した。
結果1:ミトコンドリアゲノムの全体像
まず,図1はゲノム上の遺伝子を図示したもので,当研究室で開発されたGenoMapソフトウェアにより描いてある。外側から順に右回りタンパク質遺伝
子,左回りタンパク質遺伝子,右回りRNA遺伝子,左回りRNA遺伝子を示す。イントロンはエキソンを結ぶ細い線で表した。また,イントロン内部のORF
もある。tRNA遺伝子は,チャージされるアミノ酸の一文字コードで示した。これを見ると,42個のタンパク質をコードし,その種類は被子植物のミトコン
ドリアよりも多いことがわかる。
図1の内側には,配列統計データも示した。外側から順に,GC含量(%),GC skew, AT
skewの値をそれぞれ図に示した範囲でグラフ表示している。これもGenoMapでの表示である。GC
skewは,GとCの偏りを示す数値で,ある領域に含まれるGとCの個数を使って,(G - C) / (G + C)で表される。AT
skewも同様に定義される。バクテリアゲノムでは,この値が複製開始点で反転するので,複製開始点の推定に有効とされる。ただし,シアノバクテリアなど
いくつかのゲノムでは,この手法が適用できない(おそらく複製開始点が複数あるため)。ヒメツリガネゴケに関しては,顕著な傾向は見られなかった。しか
し,遺伝子の並び方を見ると,大部分の遺伝子が右回りの鎖にコードされていることがわかる。これは,転写のしくみなどを考える上で意味ありげな結果であ
る。他の生物でも同様のことが見られる。
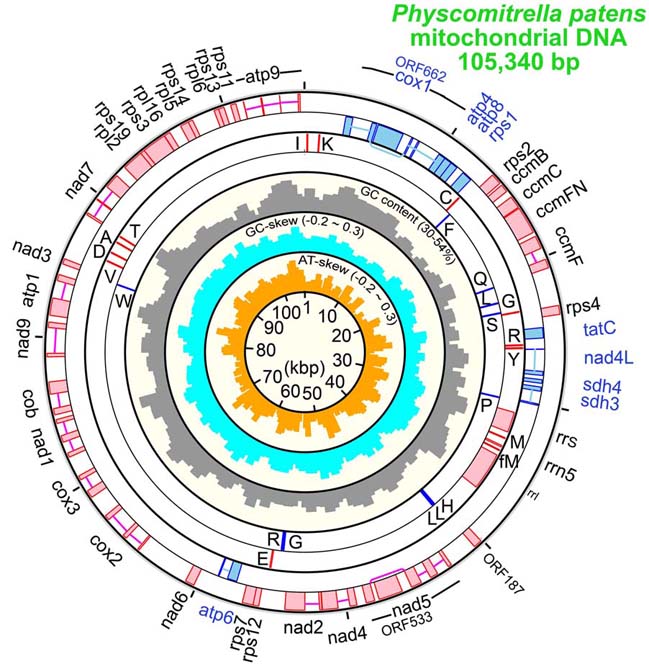
図1:ヒメツリガネゴケのミトコンドリアゲノムの構造
結果2:他の植物のミトコンドリアゲノムとの比較
さまざまな植物のミトコンドリアゲノムと,遺伝子のセットや遺伝子の並び方について,比較した。
その結果,ヒメツリガネゴケ(Physcomitrella patens),
ゼニゴケ(Marchantia polymorpha),
2種の車軸藻Chara vulgarisとChaetosphaeridium globosumの間で,遺伝子の並び
方(microsynteny)が,顕著に保存されていることがわかった。図2では遺伝子を機能別に色分けし,対応する遺伝子を線で結んだ。なお,
tRNA遺伝子は考慮していない。
一見するとごちゃごちゃのようであるが,それでも,PhyscomitrellaとCharaの間ではほとんどの遺伝子の並びが保存されていることがわかる。た
だし,水色で示したように大きな反転がある。また,PhyscomitrellaとChaetosphaeridiumとの間では遺伝子の並び方はほとんど同じ
であるが,Chaetosphaeridiumにはsdhなどいくつかの遺伝子が欠けている。また,ヒメツリガネゴケとゼニゴケ
も,いくつかのブロックの順番が異なるものの,ブロック内部での遺伝子の並び方は保存されていて,よく似ていることがわかる。
これに対し,他の緑藻や被子植物のミトコンドリアゲノムでは,リボソームタンパク質クラスターなどごく一部を除き,遺伝子の並び方はほとんど保存されてい
ない。
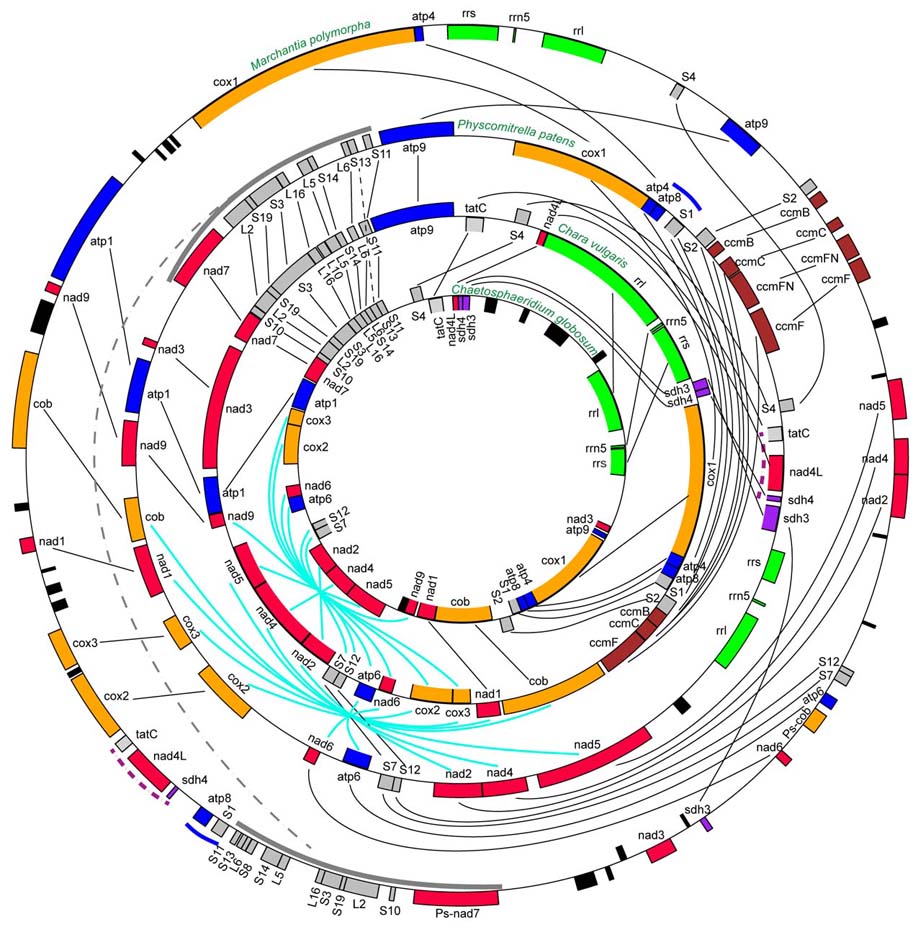
図2:ヒメツリガネゴケとゼニゴケ,2種の車軸藻との比較
結果3:系統解析
ミトコンドリアゲノムに共通にコードされた18種類のタンパク質のDNA配列の各コドンの第1,第2番目を使い,Bayesian
Inference法で系統解析を行った。
図3に結果を示す。ゼニゴケとヒメツリガネゴケが一つのグループを形成することがわかるが,他の部分の信頼度に比べ,この部分は信頼度が低く,さらに
Shimodaira-Hasegawa
(SH)テストの結果からも,両者が単系統にならずどちらかが被子植物の側系統になる系統樹を完全には排除できないことがわかった。
なお,最近,Charaの葉緑体ゲノムの解析の論文で,CharaがChaetosphaeridiumよりも根元側にある系統樹が示されている
が,それとは明確に異なる結果である。なお,この点については,SHテストの結果も明確に図3のようなトポロジーを支持している。
さらに,Mesostigmaは従来プラシノ藻として知られている
が,最近では,車軸藻の根元にくることが言われており,その考え方を支持する結果となった。

図3:ミトコンドリアゲノムにコードされた18種類のタンパク質遺伝子配列に基づく系統樹
Bayesian
Inference法で収束した結果を示す。枝の数字は収束したときのこの樹型を示す比率を示す。なお,同じデータを用いて,近隣結合法と最大節約法に
よって計算した時のブートストラップ値(パーセント)を括弧で示した。
結論
ヒメツリガネゴケのミトコンドリアゲノムは,陸上植物としてもっとも小型(105
kbp)であるが,ゼニゴケ,車軸藻などときわめてよく似た構造をもつ。被子植物では,ミトコンドリアゲノムの再編成が進み,遺伝子の並び方は全く保存さ
れていない。また,イントロンの位置についても解析したが,いくつかの遺伝子では,被子植物と共通したイントロンを持つことがわかったが,イントロンは出
入りがあるので進化の明確な指標にはならない。エディテイングは少なくとも7カ所について可能性が考えられた。今後実験的検証が必要である。コケ植物が単
系統であるかどうかについて,この結果からは明確な答えは出せない。
その他のデータ
系統解析の詳しい統計データや,イントロンの解析などは論文に出ていますので,参照してください。
GenBankのアクセッション番号は,AB251495です。
GenoMap
ソフトウェアは,当研究室ホームページからダウンロードできます。
配列統計解析は,SISEQソフト
ウェアを使いました。これも当研究室ホームページからダウンロードできます。
その他,タンパク質クラスタリングによるミトコンドリアゲノムの比較は,Gclustサーバーで提供しています。Mt23データセット
が利用できます。
Last update: Feb. 1, 2007
Copyright 2007 Sato Laboratory, University of Tokyo


